Automatisierung & KI
Automatisierung mikrobieller Analysen mit Advanced Machine Learning
Entwicklung einer fortschrittlichen KI-gestützten Lösung zur präzisen Segmentierung und automatisierten Zählung bakterieller Kolonien in mikrobiologischen Laboranalysen
Die Analyse des Bakterienwachstums auf Petrischalen ist grundlegend in der Mikrobiologie, jedoch ist das manuelle Zählen von Kolonien zeitaufwändig und mühselig. In Zusammenarbeit mit der FH Münster habe ich eine automatisierte Lösung für die Segmentierung und Zählung bakterieller Kolonien entwickelt, welche den Analyseprozess deutlich beschleunigt.
Aufbau einer effizienten Datenpipeline
Um den Zählprozess zu automatisieren, haben wir zunächst eine spezielle Fotoapparatur verwendet, die hochwertige Bilder von Petrischalen mit Bakterienkulturen aufnimmt. Ausgestattet mit einer Raspberry Pi High Quality Camera erzeugt dieses Setup Bilder mit 9,24 Megapixeln, welche eine gute Basis für das Training von Machine-Learning-Modellen bilden.
Effiziente Datenannotation mit KI-Unterstützung
Für die effiziente Annotation der Daten entwickelte ich eine eigene Webanwendung, die das Segment Anything Model (SAM) integriert. Dies ermöglichte es mir und den Kolleg*innen der FH Münster, schnell und präzise Segmentierungsmasken durch einfache Klick-Interaktionen zu erstellen und den Aufwand bei der Annotation erheblich zu reduzieren. Auf diese Weise entstand ein Datensatz aus 200 präzise annotierten Bildern.
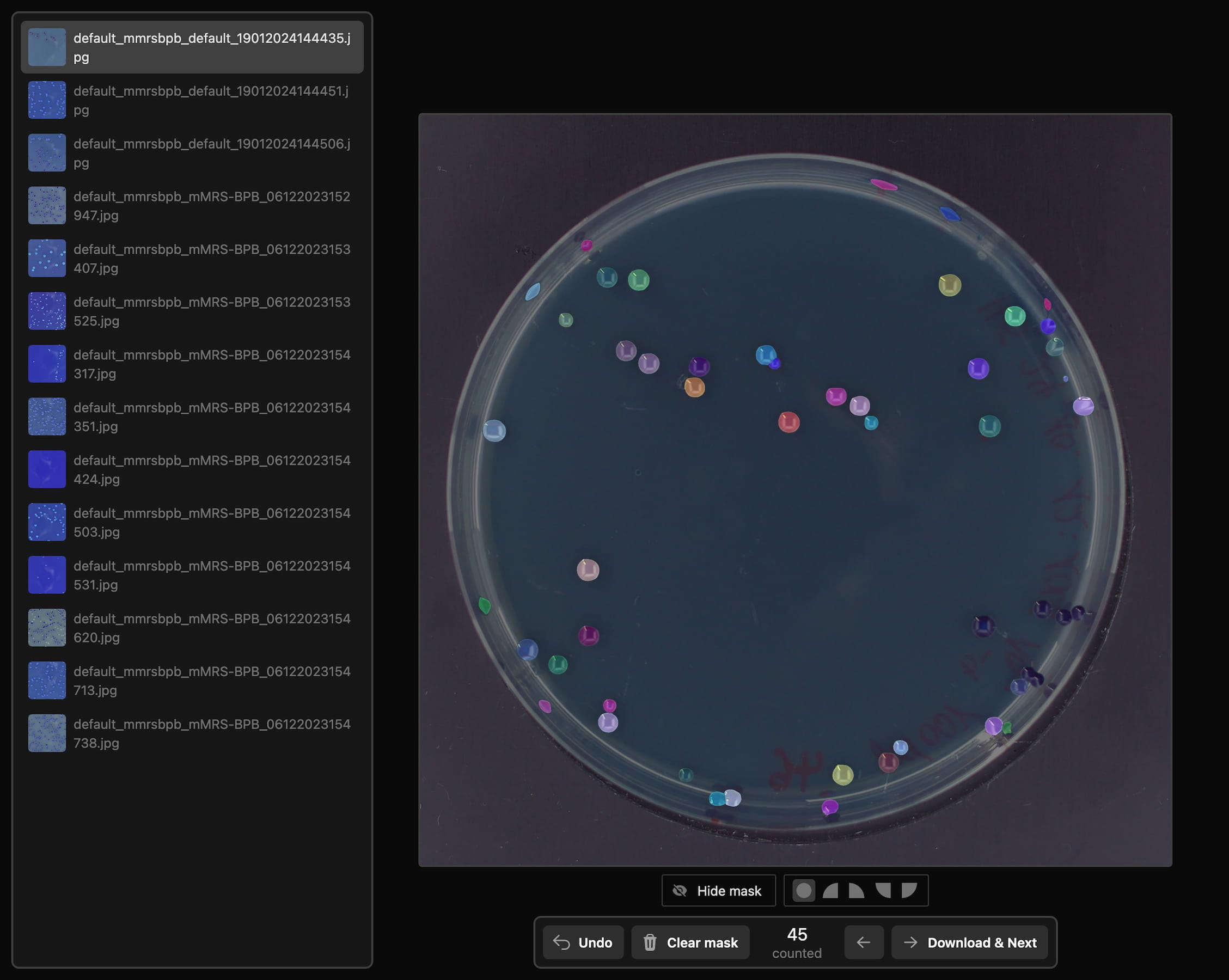
Präzise Instance-Segmentation-Masken, erstellt mithilfe der SAM-basierten Annotation.
Optimierung der Genauigkeit durch strategisches Training
Als Erkennungsmodell habe ich die Faster R-CNN Modellarchitektur eingesetzt, welche für ihre hohe Genauigkeit bei Objekterkennungsaufgaben bekannt ist. Ich habe das Modell strategisch zunächst auf dem COCO-Datensatz vortrainiert und anschließend mit dem spezialisierten AGAR-Datensatz finetuned, bevor das Modell mit dem eigen-gesammelten Dataset final trainiert wurde. Dieses Vorgehen verbesserte die Leistung des Modells deutlich, wodurch eine hohe Präzision und niedrige Fehlerquote erreicht wurde.
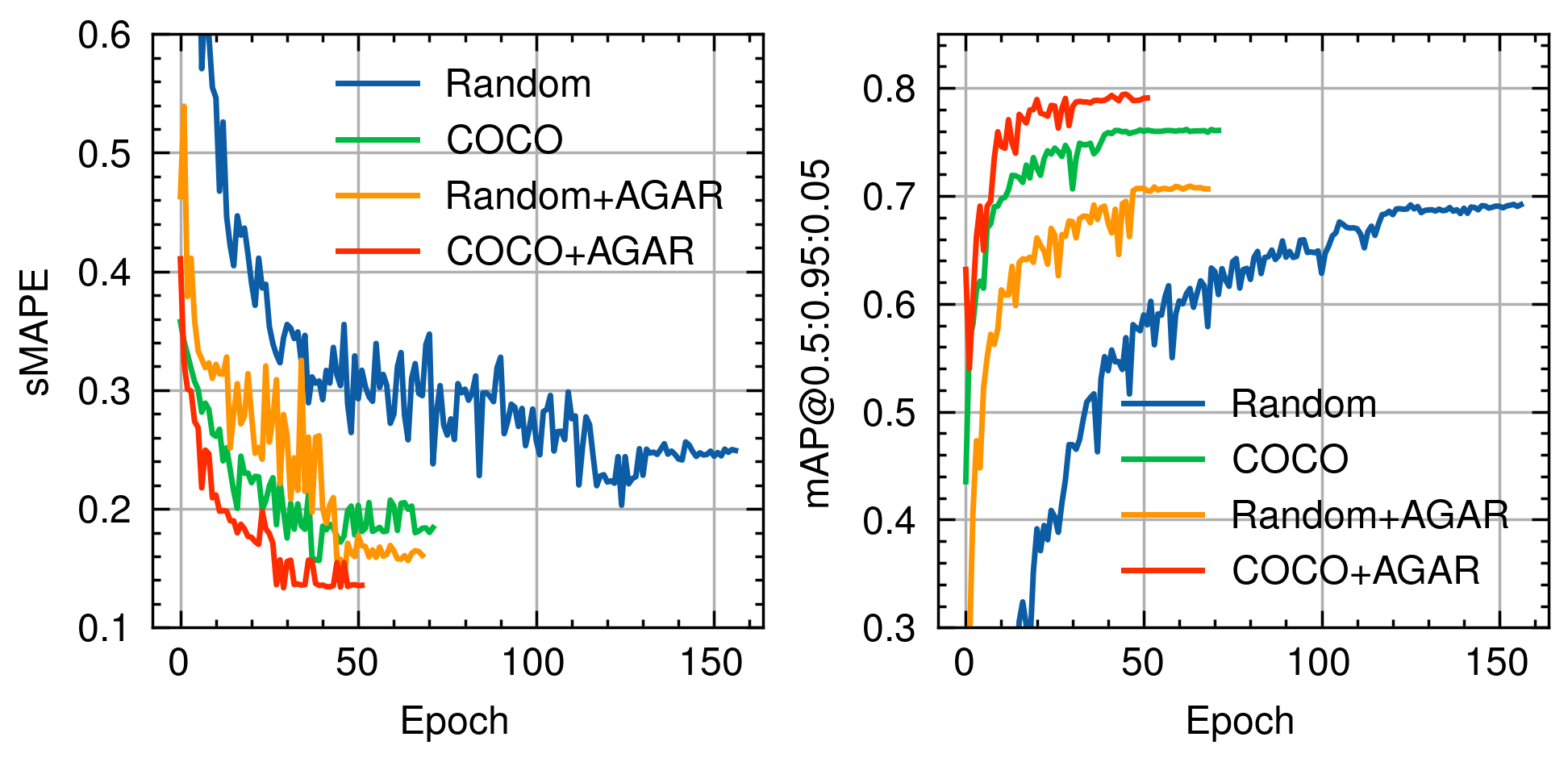
Verbesserung der Validierungsleistung durch strategisches Vortraining.
Schnelle Koloniezählung
Mein trainiertes Faster R-CNN Modell erzielt bemerkenswerte Inferenzgeschwindigkeiten, indem es bakterielle Kolonien in etwa vier Sekunden auf einem herkömmlichen Computer erkennt. Diese automatisierte Zählmethode beschleunigt den mikrobiologischen Analyseprozess enorm, welcher bei manuellem Zählen mehrere Minuten in Anspruch nehmen kann.
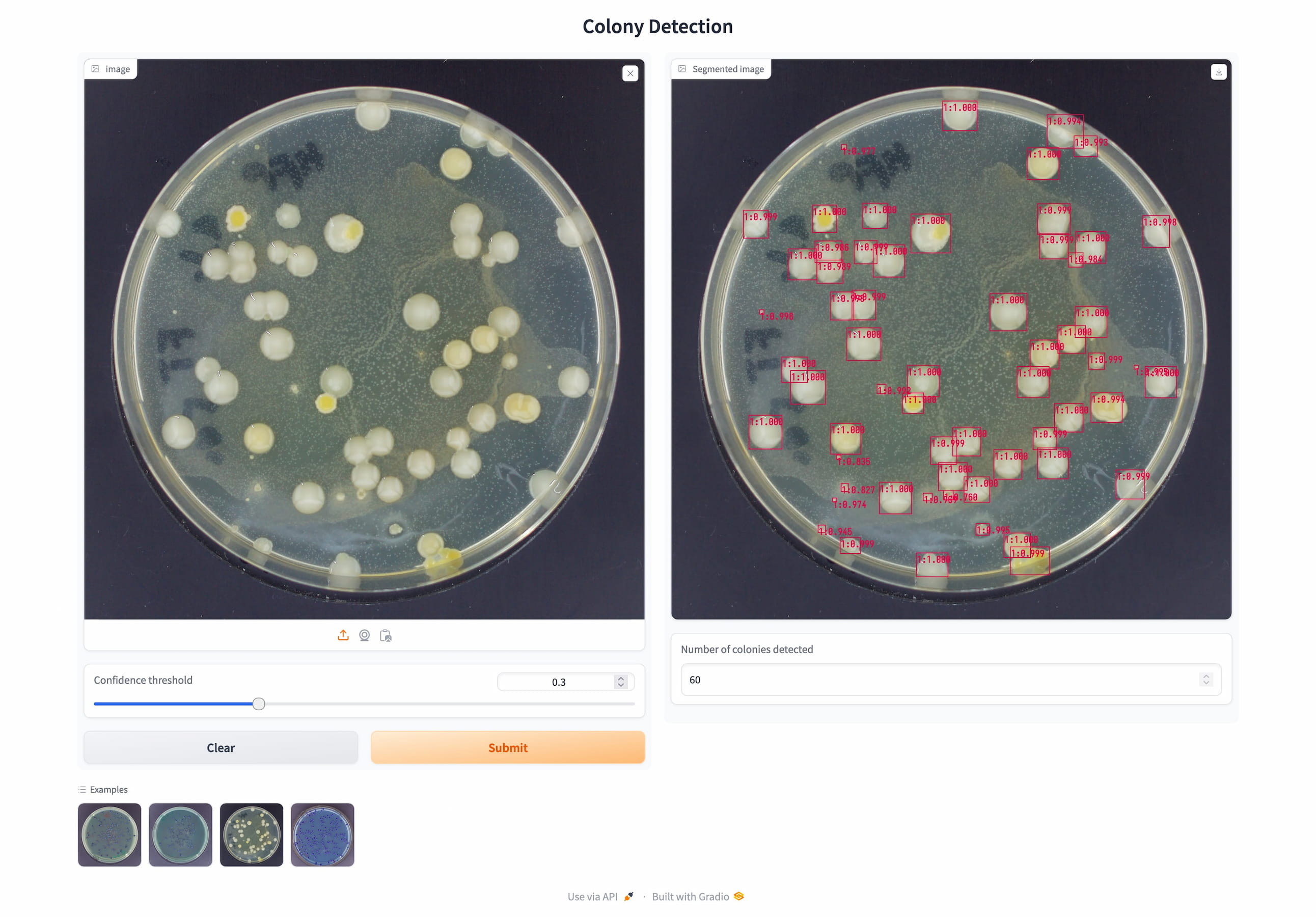
Benutzerfreundliche Inferenzanwendung, die an der FH Münster für schnelle Kolonieerkennung eingesetzt wird.
Robuste und skalierbare Erkennung
Die quantitative Bewertung meines Modells verdeutlicht dessen Robustheit:
Die meisten Erkennungen stellen tatsächlich bakterielle Kolonien dar.
Das Modell erkennt die überwiegende Mehrheit der vorhandenen Kolonien.
Mittlerer absoluter Fehler von nur 6,26 Kolonien pro Platte im Durchschnitt (von teilweise hunderten Kolonien pro Platte).
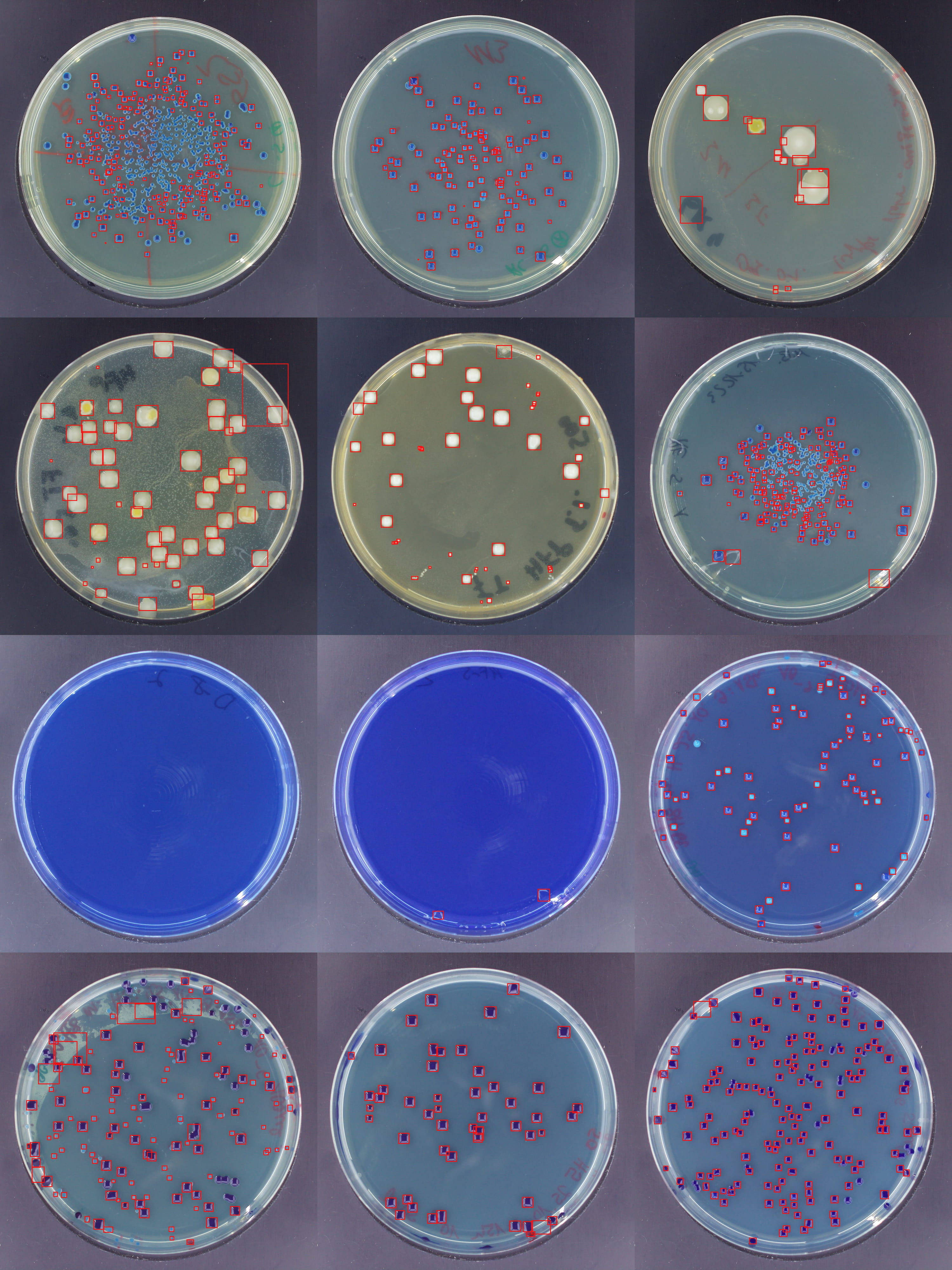
Beispiele für automatische Erkennungen, welche die Fähigkeit des Modells zur Verarbeitung verschiedener Kolonieformen und -dichten zeigen.
Praktische Anwendung und zukünftige Skalierbarkeit
Um eine hohe Praxistauglichkeit zu gewährleisten, habe ich mein Modell in die oben gezeigte benutzerfreundliche Webanwendung integriert, die an der FH Münster eingesetzt wird, um Forschenden eine schnelle Automatisierung der Koloniezählung zu ermöglichen. Das System ist für Skalierbarkeit ausgelegt, wobei fortlaufende Verbesserungen durch die Ergänzung weiterer annotierter Daten erfolgen können.
Dieses Projekt demonstriert meine Expertise im Bereich Machine Learning und Automatisierung und bietet eine praktische und effiziente Lösung für mikrobiologische Analysen.